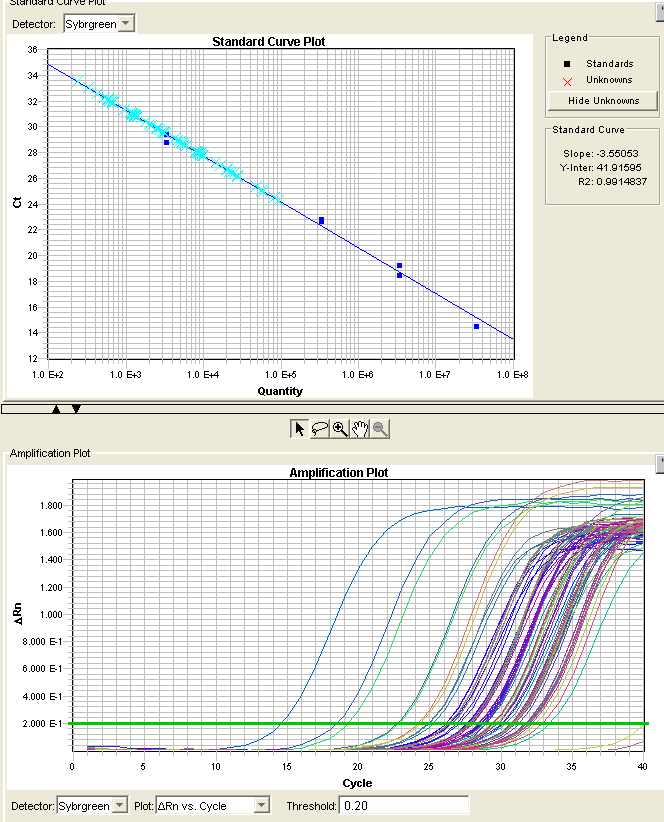
le graphe du dessus c& #39;est la relation entre copies et Ct. Les points bleus pleins ce sont les points de la gamme de référence. Donc on une solution avec un titre connu, et on passe plusieurs concentrations.
le point en bas à droite a une valeur à 2.5.e7 copies et on voit que le Ct calculé est à 15. En regardant le graphe Amplification plot, on voit que c& #39;est la courbe bleue à gauche et qu& #39;il y a une ligne verte qui définit le seuil pour le choix du Ct 3/x
un Ct d& #39;écart, c& #39;est en théorie une multiplication par 2, en pratique 1.8, donc 2 Ct d& #39;écart c& #39;est 3.24 fois moins de matrices entre deux échantillons. 4/4
Ah si on voit que deux échantillons ont des débuts à un Ct34 ou 38 . Sont-ce des fragments d& #39;ADN morts...non juste qu& #39;on a peu-être une petite contamination inter-puits, une micro-goutte lors du prélèvement qui a contaminé ce puit avec de très faibles quantités ou 5/x
ou alors de l& #39;hybridation aspécifique, car les primers peuvent d& #39;associer entre eux et donner in fine un signal, car ce qu& #39;on mesure c& #39;est une émission sous UV. Et comment faire pour savoir si on a des dimères de primer, et bien on fait une melt curve 6/x
là un test de différent primer à 2 températures d& #39;hybridation, 60°C au dessus et 55°C en dessous. La présence de dimères est mesurée par l& #39;apparition de pics à des températures plus faible (vers 70°C) alors que le fragment amplifié qui est de plus grande taille a un pic à 90° 7/x
donc en l& #39;absence de matrice, l& #39;amplification de dimères peut être favorisé même si on sélectionne des primers qui ne doivent pas faire de dimères en théorie. C& #39;est la courbe T- qui correspond au mélange sans la matrice ADN et juste les primers. On voit le pic parfois juste 8/x
dans le témoin (panel bas couple 806R/516F R pour reverse et F pour forward) et parfois même avec la matrice (pas bon car cela amène à sur-estimer le nombre de copies). Donc dans notre cas on sélectionne le couple 806R/516F avec une température d& #39;hybridation à 55°C) 9/x
Donc quand quelqu& #39;un vous dit qu& #39;au dessus d& #39;une valeur de Ct on ne mesure que des virus morts, non elle réfléchit à l& #39;envers. Si le travail est bien fait, les primers bien paramétrés, les melting curves connues, on peut avoir un résultat fiable. 10/x
Merci à Julie, Mélanie et Nicols qui ont fait les manips que je vous ai montré (c& #39;est de la vraie science). Promis on vous parlera des couples utilisés pour les Q-RTPCR du covid, comment savoir si les virus sont vivants et virulents (deux choses différentes, un virus muté 11/x
pour ne plus infecter une cellule humaine est bien formé mais non virulent par exemple. poke @monox @DavidPatsouris pour le travail collectif

 Read on Twitter
Read on Twitter