Une nouvelle étape vers la stérilité mâle génique à façon chez les plantes cultivées grâce à l& #39;édition du génome via Crispr/Cas9
Et si demain l’édition génomique permettait de produire des hybrides F1 plus simplement et pour moins cher ?
 https://abs.twimg.com/emoji/v2/... draggable="false" alt="⬇️" title="Pfeil nach unten" aria-label="Emoji: Pfeil nach unten">
https://abs.twimg.com/emoji/v2/... draggable="false" alt="⬇️" title="Pfeil nach unten" aria-label="Emoji: Pfeil nach unten"> https://abs.twimg.com/emoji/v2/... draggable="false" alt="⬇️" title="Pfeil nach unten" aria-label="Emoji: Pfeil nach unten">
https://abs.twimg.com/emoji/v2/... draggable="false" alt="⬇️" title="Pfeil nach unten" aria-label="Emoji: Pfeil nach unten"> https://abs.twimg.com/emoji/v2/... draggable="false" alt="⬇️" title="Pfeil nach unten" aria-label="Emoji: Pfeil nach unten">
https://abs.twimg.com/emoji/v2/... draggable="false" alt="⬇️" title="Pfeil nach unten" aria-label="Emoji: Pfeil nach unten">
Et si demain l’édition génomique permettait de produire des hybrides F1 plus simplement et pour moins cher ?
Aujourd’hui ¾ des surfaces de production de semences hybrides de maïs nécessitent la castration manuelle et/ou mécanique entrainant des couts importants et des contraintes non négligeables en termes de main d’œuvre (et aggravées en ces temps de COVID19).
Un système efficace et facile a mettre en place de castration génétique pourrait réduire ces couts et ces contraintes
L& #39;étude est ici
https://doi.org/10.1016/j.molp.2020.06.003">https://doi.org/10.1016/j...
L& #39;étude est ici
https://doi.org/10.1016/j.molp.2020.06.003">https://doi.org/10.1016/j...
La stérilité mâle génique est un processus qui est largement utilisé pour la production d& #39;hybrides F1 en agriculture
Si vous devez vous mettre à jour sur "pourquoi les hybrides F1" c& #39;est par là https://twitter.com/QADPlantes/status/1266123891665178624?s=20">https://twitter.com/QADPlante...
Si vous devez vous mettre à jour sur "pourquoi les hybrides F1" c& #39;est par là https://twitter.com/QADPlantes/status/1266123891665178624?s=20">https://twitter.com/QADPlante...
Et si vous devez vous mettre à jour sur "comment les hybrides F1" et plus particulièrement la stérilité mâle génique (SMG) c& #39;est par ici https://twitter.com/QADPlantes/status/1266128968333373440?s=20">https://twitter.com/QADPlante...
Je recommande fortement la lecture de ces fils au risque d’être un peu perdu par la suite
Les technologies de contrôle de l& #39;hybridation (Stérilité Male Génique [SMG] et Stérilité Mâle cytoplasmique [SMC] ont chacune leurs avantages et leur inconvénients. Les SMC sont dépendantes de l& #39;identification de gènes de stérilité mitochondriaux dans des
populations sauvages (pour faire un raccourci) et les SMG nécessitent de travailler avec des allèles récessifs et d& #39;effectuer un tri lors de la production du parent femelle de l& #39;hybride F1 (1 graine sur 2 à conserver seulement)
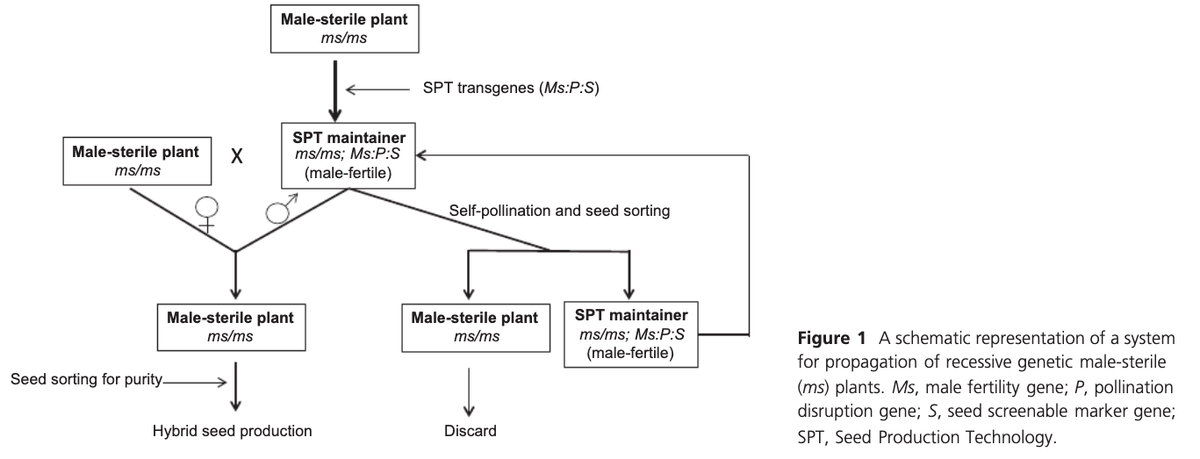
Récemment de nouvelles stratégies de SMG ont été proposées : parmi elles on peut citer la SPT (Seed Production Technology)
https://onlinelibrary.wiley.com/doi/epdf/10.1111/pbi.12477">https://onlinelibrary.wiley.com/doi/epdf/...
https://onlinelibrary.wiley.com/doi/epdf/10.1111/pbi.12477">https://onlinelibrary.wiley.com/doi/epdf/...
Elle est constituée de 3 composants :
1- une restauration de la fertilité par un transgène (MS sur le schéma)
2- l& #39;inactivation du développement du pollen portant le transgène (P sur le schéma)
3- un marqueur phénotypique pour le tri sur graine du mainteneur (S sur le schéma)
1- une restauration de la fertilité par un transgène (MS sur le schéma)
2- l& #39;inactivation du développement du pollen portant le transgène (P sur le schéma)
3- un marqueur phénotypique pour le tri sur graine du mainteneur (S sur le schéma)
Ce système permet de transformer une plante mâle stérile en parent mainteneur (fertile) ET incapable de transmettre cette fertilité transgénique par la voie mâle (la moitié des grains de pollen sont porteurs de P et ne participent pas à la fécondation).
Le mainteneur est trié directement sur graine pour faciliter l& #39;utilisation à la prochaine génération. Ce système permet de produire un parent femelle non transgénique de l& #39;hybride F1.
Ce système nécessite toutefois de disposer au préalable une lignée SMG, qui est souvent un mutant de fertilité (ms/ms), et le processus est toujours autant tributaire des nombreux back cross nécessaires entre lignées lors de l& #39;établissement du couple SMG/mainteneur.
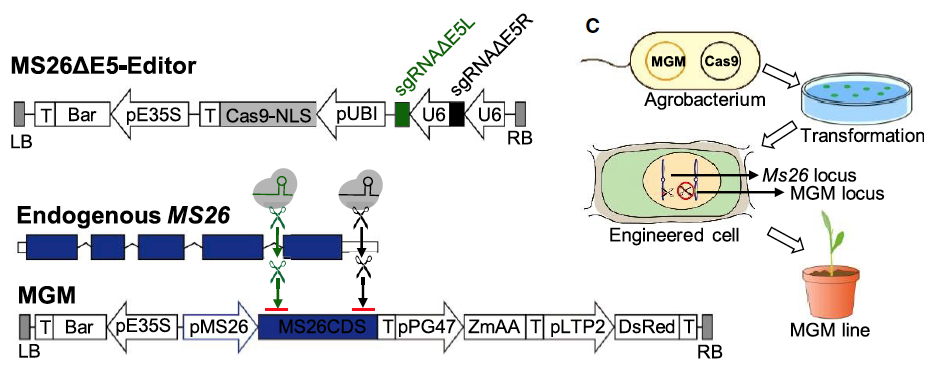
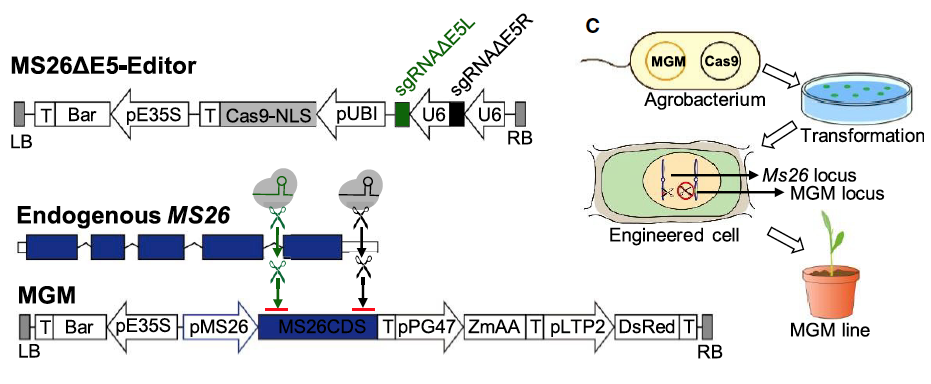
L& #39;étude présente ici la création par édition génomique d& #39;une lignée SMG (à partir d& #39;une lignée fertile quelconque) et d& #39;un mainteneur associé pour la production de semences hybrides.
Le design est le suivant :
Créer par Crispr en une étape une lignée a la fois :
- SMG par disruption d& #39;un gène de fertilité, ici ZmMS26 (MS26DE5 sur le schéma)
Créer par Crispr en une étape une lignée a la fois :
- SMG par disruption d& #39;un gène de fertilité, ici ZmMS26 (MS26DE5 sur le schéma)
- mainteneur (appelé MGM) par l& #39;expression des 3 modules présentés dans le système SPT
 https://abs.twimg.com/emoji/v2/... draggable="false" alt="1⃣" title="Tastenkappe Ziffer 1" aria-label="Emoji: Tastenkappe Ziffer 1"> la séquence codante de ZmMS26 pour restaurer la fertilité (MS26CDS)
https://abs.twimg.com/emoji/v2/... draggable="false" alt="1⃣" title="Tastenkappe Ziffer 1" aria-label="Emoji: Tastenkappe Ziffer 1"> la séquence codante de ZmMS26 pour restaurer la fertilité (MS26CDS)
 https://abs.twimg.com/emoji/v2/... draggable="false" alt="2⃣" title="Tastenkappe Ziffer 2" aria-label="Emoji: Tastenkappe Ziffer 2">l& #39;inactivateur de développement du pollen (pPG47-ZmAA)
https://abs.twimg.com/emoji/v2/... draggable="false" alt="2⃣" title="Tastenkappe Ziffer 2" aria-label="Emoji: Tastenkappe Ziffer 2">l& #39;inactivateur de développement du pollen (pPG47-ZmAA)
 https://abs.twimg.com/emoji/v2/... draggable="false" alt="3⃣" title="Tastenkappe Ziffer 3" aria-label="Emoji: Tastenkappe Ziffer 3"> le marqueur phénotypique ici fluorescent (pLTP2-DsRed)
https://abs.twimg.com/emoji/v2/... draggable="false" alt="3⃣" title="Tastenkappe Ziffer 3" aria-label="Emoji: Tastenkappe Ziffer 3"> le marqueur phénotypique ici fluorescent (pLTP2-DsRed)
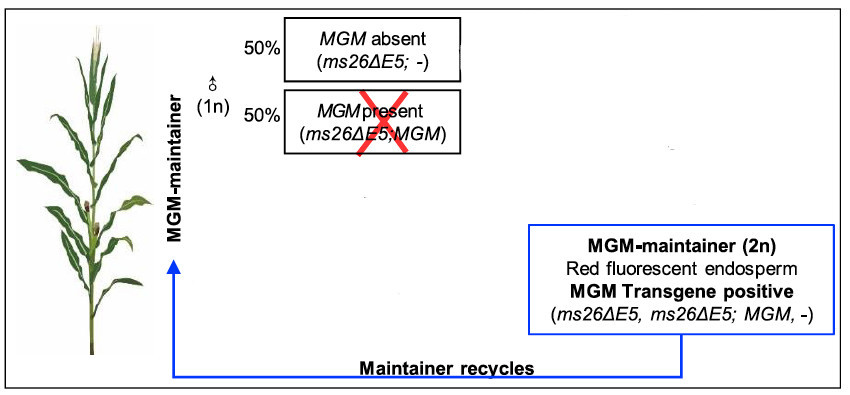
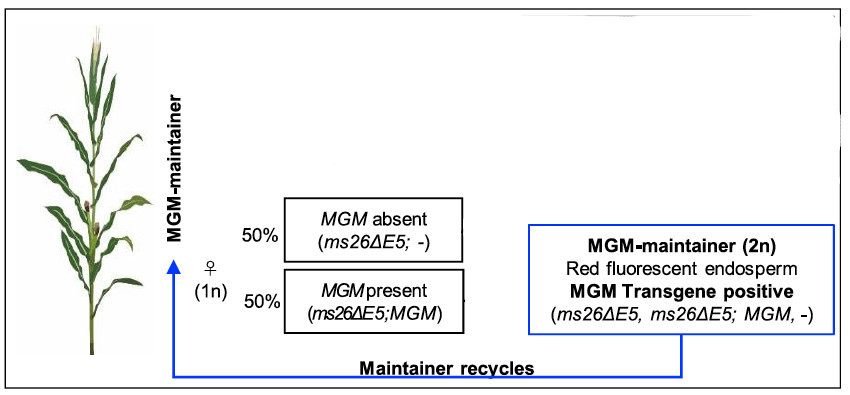
Cette lignée sera donc fertile et regardons ce qui se passera au moment de la production des gamètes :
Coté mâle : seuls les gamètes mâles dépourvus de transgène sont viables et transmette la disruption du gène ZmMS26
Coté mâle : seuls les gamètes mâles dépourvus de transgène sont viables et transmette la disruption du gène ZmMS26
Coté femelle : tous les gamètes transmettent la disruption du gène ZmMS26 et la moitié transmettent en plus le fragment MGM
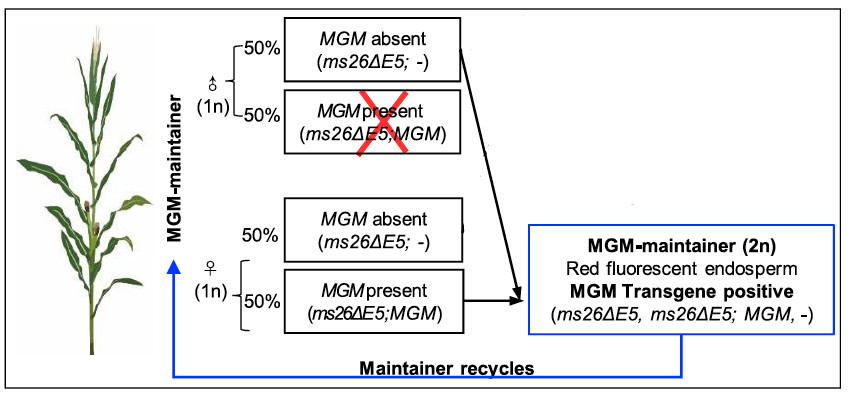
La fécondation va donc mettre en jeu 1 seul type de gamètes mâles et 2 types de gamètes femelles, ce qui produira 2 types de plantes (important pour la suite)
Les mainteneurs :
Fertiles, issus de graines portant le marqueur phénotypique (Red fluorescent) et homozygotes pour la disruption du gène ZmMS26
Fertiles, issus de graines portant le marqueur phénotypique (Red fluorescent) et homozygotes pour la disruption du gène ZmMS26
Les mâle-stériles :
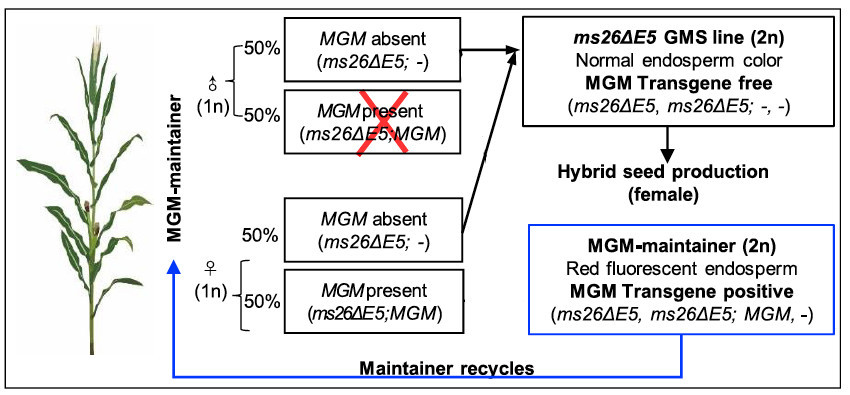
homozygotes pour la disruption du gène ZmMS26 mais exempts du fragment MGM (donc non transgéniques) et ne portant pas le marqueur phénotypique. Ce seront les parents femelles de la production d& #39;hybride (ceux qu& #39;on castre auj. a la main ou a la machine)
homozygotes pour la disruption du gène ZmMS26 mais exempts du fragment MGM (donc non transgéniques) et ne portant pas le marqueur phénotypique. Ce seront les parents femelles de la production d& #39;hybride (ceux qu& #39;on castre auj. a la main ou a la machine)
En triant sur la présence/absence du marqueur phénotypique on a donc potentiellement un moyen utilisable à haut débit de distinguer les 2 types directement sur graine.
Quels sont les résultats des auteurs ?
Quels sont les résultats des auteurs ?
Je passe sur l& #39;obtention des lignées SMG, procédure assez classique d& #39;obtention et de caractérisation d& #39;une délétion par édition du génome. Les auteurs ont sélectionné les lignées homozygotes pour la disruption du gène ZmMS26 et
ayant à la fois ségrégé le MS26DE5-Editor (plus nécessaire vu le statut du gène ZmMS26) et comportant le fragment MGM en 1 seule copie (la transgenèse pouvant générer des copies multiples dans le génome).
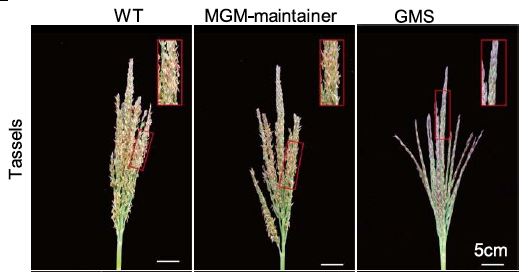
Les auteurs ont caractérisé les plantes SMG et MGM issues d& #39;une lignée choisie et ont caractérisé différents critères :
- leur morphologie globale (WT = lignée de départ)
- leur morphologie globale (WT = lignée de départ)
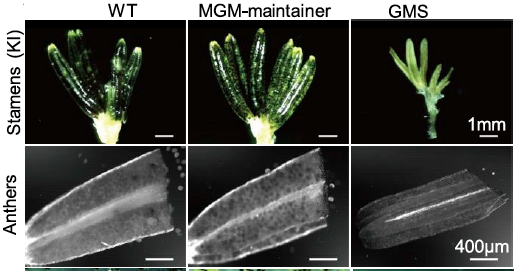
- Les étamines et les anthères qui portent les gamètes mâles (anormales chez la lignée SMG mais pas chez la lignée MGM comme attendu)
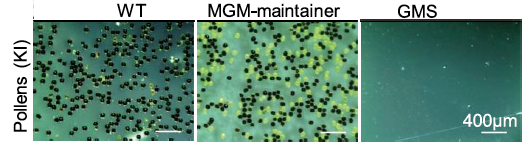
La viabilité du pollen (violet = viable , jaune = mort). Ils ont confirmé la proportion 50-50 viable/mort comme attendu dans la ligné MGM et l& #39;absence de pollen dans la lignée SMG (comme attendu ... revenez en arrière si vous ne comprenez pas pourquoi)
Ces résultats confirment que la disruption du gène ZmMS26 confère bien la SMG. De plus ces analyses suggèrent la fonctionnalité du module MGM même si je regrette que les auteurs ne montrent pas formellement l& #39;absence de sa transmission a la descendance sur plusieurs générations.
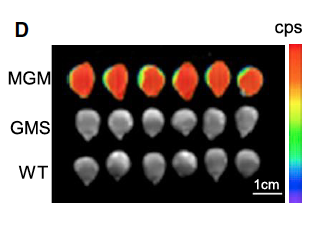
Marqueur phénotypique sur graines
Le mainteneur MGM possède la caractéristique d& #39;exprimer dans ses graines un marqueur permettant de le différencier d& #39;une lignée SMG
Le mainteneur MGM possède la caractéristique d& #39;exprimer dans ses graines un marqueur permettant de le différencier d& #39;une lignée SMG
Les auteurs ont examiné la capacité à trier les grains directement sur un épi issu d& #39;une autofécondation sous une lampe ad hoc. Les grains porteurs du marqueur fluorescent en rouge (et donc porteurs du fragment MGM) là où les graines qui donneront des plantes SMG restent sombres
Cette analyse non destructive permettrait même un tri à l& #39;aide d’un trieur automatique par la mesure de l& #39;intensité de la fluorescence (cps)
Cet article de "preuve de concept" du couplage entre l& #39;utilisation de CrisprCas9 et de la technologie de type SPT pourrait a terme permettre le développement de lignées permettant la production facilité et a moindre cout des parents des hybrides F1 semés par les agriculteurs

 Read on Twitter
Read on Twitter https://abs.twimg.com/emoji/v2/... draggable="false" alt="⬇️" title="Pfeil nach unten" aria-label="Emoji: Pfeil nach unten">https://abs.twimg.com/emoji/v2/... draggable="false" alt="⬇️" title="Pfeil nach unten" aria-label="Emoji: Pfeil nach unten">" title="Une nouvelle étape vers la stérilité mâle génique à façon chez les plantes cultivées grâce à l& #39;édition du génome via Crispr/Cas9Et si demain l’édition génomique permettait de produire des hybrides F1 plus simplement et pour moins cher ?https://abs.twimg.com/emoji/v2/... draggable="false" alt="⬇️" title="Pfeil nach unten" aria-label="Emoji: Pfeil nach unten">https://abs.twimg.com/emoji/v2/... draggable="false" alt="⬇️" title="Pfeil nach unten" aria-label="Emoji: Pfeil nach unten">https://abs.twimg.com/emoji/v2/... draggable="false" alt="⬇️" title="Pfeil nach unten" aria-label="Emoji: Pfeil nach unten">" class="img-responsive" style="max-width:100%;"/>
https://abs.twimg.com/emoji/v2/... draggable="false" alt="⬇️" title="Pfeil nach unten" aria-label="Emoji: Pfeil nach unten">https://abs.twimg.com/emoji/v2/... draggable="false" alt="⬇️" title="Pfeil nach unten" aria-label="Emoji: Pfeil nach unten">" title="Une nouvelle étape vers la stérilité mâle génique à façon chez les plantes cultivées grâce à l& #39;édition du génome via Crispr/Cas9Et si demain l’édition génomique permettait de produire des hybrides F1 plus simplement et pour moins cher ?https://abs.twimg.com/emoji/v2/... draggable="false" alt="⬇️" title="Pfeil nach unten" aria-label="Emoji: Pfeil nach unten">https://abs.twimg.com/emoji/v2/... draggable="false" alt="⬇️" title="Pfeil nach unten" aria-label="Emoji: Pfeil nach unten">https://abs.twimg.com/emoji/v2/... draggable="false" alt="⬇️" title="Pfeil nach unten" aria-label="Emoji: Pfeil nach unten">" class="img-responsive" style="max-width:100%;"/>
 la séquence codante de ZmMS26 pour restaurer la fertilité (MS26CDS)https://abs.twimg.com/emoji/v2/... draggable="false" alt="2⃣" title="Tastenkappe Ziffer 2" aria-label="Emoji: Tastenkappe Ziffer 2">l& #39;inactivateur de développement du pollen (pPG47-ZmAA)https://abs.twimg.com/emoji/v2/... draggable="false" alt="3⃣" title="Tastenkappe Ziffer 3" aria-label="Emoji: Tastenkappe Ziffer 3"> le marqueur phénotypique ici fluorescent (pLTP2-DsRed)" title="- mainteneur (appelé MGM) par l& #39;expression des 3 modules présentés dans le système SPThttps://abs.twimg.com/emoji/v2/... draggable="false" alt="1⃣" title="Tastenkappe Ziffer 1" aria-label="Emoji: Tastenkappe Ziffer 1"> la séquence codante de ZmMS26 pour restaurer la fertilité (MS26CDS)https://abs.twimg.com/emoji/v2/... draggable="false" alt="2⃣" title="Tastenkappe Ziffer 2" aria-label="Emoji: Tastenkappe Ziffer 2">l& #39;inactivateur de développement du pollen (pPG47-ZmAA)https://abs.twimg.com/emoji/v2/... draggable="false" alt="3⃣" title="Tastenkappe Ziffer 3" aria-label="Emoji: Tastenkappe Ziffer 3"> le marqueur phénotypique ici fluorescent (pLTP2-DsRed)" class="img-responsive" style="max-width:100%;"/>
la séquence codante de ZmMS26 pour restaurer la fertilité (MS26CDS)https://abs.twimg.com/emoji/v2/... draggable="false" alt="2⃣" title="Tastenkappe Ziffer 2" aria-label="Emoji: Tastenkappe Ziffer 2">l& #39;inactivateur de développement du pollen (pPG47-ZmAA)https://abs.twimg.com/emoji/v2/... draggable="false" alt="3⃣" title="Tastenkappe Ziffer 3" aria-label="Emoji: Tastenkappe Ziffer 3"> le marqueur phénotypique ici fluorescent (pLTP2-DsRed)" title="- mainteneur (appelé MGM) par l& #39;expression des 3 modules présentés dans le système SPThttps://abs.twimg.com/emoji/v2/... draggable="false" alt="1⃣" title="Tastenkappe Ziffer 1" aria-label="Emoji: Tastenkappe Ziffer 1"> la séquence codante de ZmMS26 pour restaurer la fertilité (MS26CDS)https://abs.twimg.com/emoji/v2/... draggable="false" alt="2⃣" title="Tastenkappe Ziffer 2" aria-label="Emoji: Tastenkappe Ziffer 2">l& #39;inactivateur de développement du pollen (pPG47-ZmAA)https://abs.twimg.com/emoji/v2/... draggable="false" alt="3⃣" title="Tastenkappe Ziffer 3" aria-label="Emoji: Tastenkappe Ziffer 3"> le marqueur phénotypique ici fluorescent (pLTP2-DsRed)" class="img-responsive" style="max-width:100%;"/>